首页 > 医疗资讯/ 正文
普拉替尼等选择性RET抑制剂已成为RET融合阳性非小细胞肺癌(NSCLC)患者的标准治疗方案。循环肿瘤DNA(ctDNA)动态监测已被证实可有效评估NSCLC患者的病情控制与进展情况及治疗应答效果。本前瞻性研究纳入接受普拉替尼治疗的中国晚期RET融合阳性NSCLC患者,采用等位基因频率相关指标、游离DNA(cfDNA)定量标准化指标及甲基化相关指标,分析其治疗基线、治疗第8周及疾病进展时的ctDNA纵向变化特征(临床试验注册号:NCT03037385),并探究ctDNA动态变化与肿瘤应答及基因组变异之间的关联。
研究共纳入21例患者,结果显示,基线时存在PIK3CA共突变的患者无进展生存期更短(PFS;3.0个月vs 12.4个月,P<0.001)。在所有检测指标中,基线ctDNA水平较低的患者均表现出更长的PFS:等位基因频率相关指标组的HR为0.24(95%CI为0.07 - 0.80,P=0.012);cfDNA定量标准化指标组的HR为0.20(95%CI为0.05 - 0.71,P=0.006);甲基化相关指标组的HR为0.09(95%CI为0.01 - 0.85,P=0.010)。首次影像学评估时ctDNA早期清除的患者,其PFS同样显著延长(中位PFS未达到vs 4.8个月,P=0.002),且疾病控制率更高(71.4%vs 0%)。此外,研究发现ctDNA存在三种不同的动态变化模式,即清除 - 回升型、下降 - 回升型和持续清除型,这些模式分别对应不同的疾病进展特征,包括全身性进展、新增胸腔外病灶以及新增颅内或胸腔内病灶。疾病进展时未发现组织学转化的相关证据。ctDNA回升样本中出现了KRAS G12R突变及其他非经典变异。分子层面的疾病进展较影像学所示的进展平均提前2.2个月。上述研究结果表明,采用多种指标进行ctDNA监测,可早期预测RET融合阳性NSCLC患者的肿瘤治疗应答情况与疾病进展趋势。ctDNA早期清除现象及其动态变化模式可作为指导临床早期干预的无创生物标志物,基于ctDNA制定的治疗策略值得进一步验证。
RET融合在1%-2%的晚期NSCLC患者中被检出,此类患者可采用普拉替尼等高效抑制剂进行靶向治疗。但目前,如何早期筛选治疗有效人群、及时识别耐药情况仍面临挑战,且耐药机制尚未完全明确。以ctDNA为代表的液体活检生物标志物,为无创、动态监测疾病病程提供了可能。既往研究已证实,通过分析血浆cfDNA,能够监测可靶向的RET变异位点,同时也可评估治疗启动后ctDNA的清除情况。不过,这些研究结论仍需经随访时间更长、ctDNA动态特征描述更详尽的研究进一步验证。此外,目前已提出多种基于ctDNA的检测指标,但这些指标的应用效能仍有待深入评估。
本研究对21例接受普拉替尼治疗的中国NSCLC患者前瞻性队列展开探索性分析(临床试验注册号:NCT03037385)。该分析以母研究的主要终点客观缓解率(ORR),以及母研究中的PFS数据为依据。分别在治疗基线、治疗第8周及疾病进展时采集患者外周血。本研究计算了三项cfDNA相关指标,分别为变异等位基因频率峰值(maxAF)、游离DNA定量标准化后的平均肿瘤分子数(MTM/mL)以及基于甲基化的恶性度密度(MD)比值。本研究分析了临床结局与ctDNA动态变化的相关性,并通过cfDNA基因组分析,明确了普拉替尼可能存在的继发性耐药机制。本研究的中位随访时间为17.5个月(95%CI为12.1 - 21.8个月)。
治疗前ctDNA特征与普拉替尼治疗结局的相关性:
2例存在PIK3CA共突变(具体突变类型为E545K和H1047L)的患者,其PFS显著短于无该共突变的患者(中位PFS:3.0个月vs 12.4个月,P<0.001,见图1a)。多因素分析进一步证实,该关联独立于治疗线数。在所有ctDNA检测指标中,治疗前ctDNA水平较高的患者PFS均更短 (maxAF: HR = 0.24,P = 0.012;MTM/mL:HR = 0.20,P = 0.006;MD比值:HR = 0.09,P = 0.010;图1b-d)。
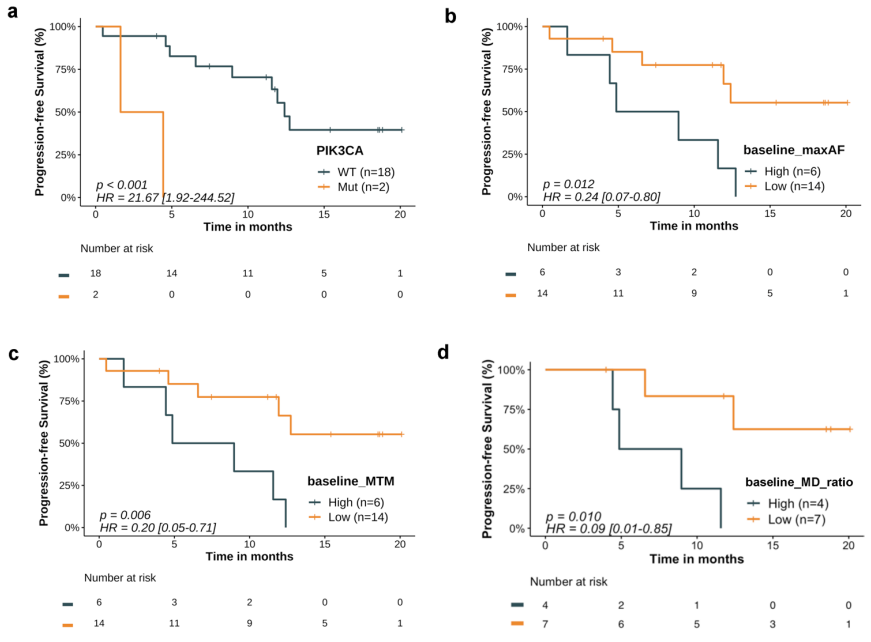
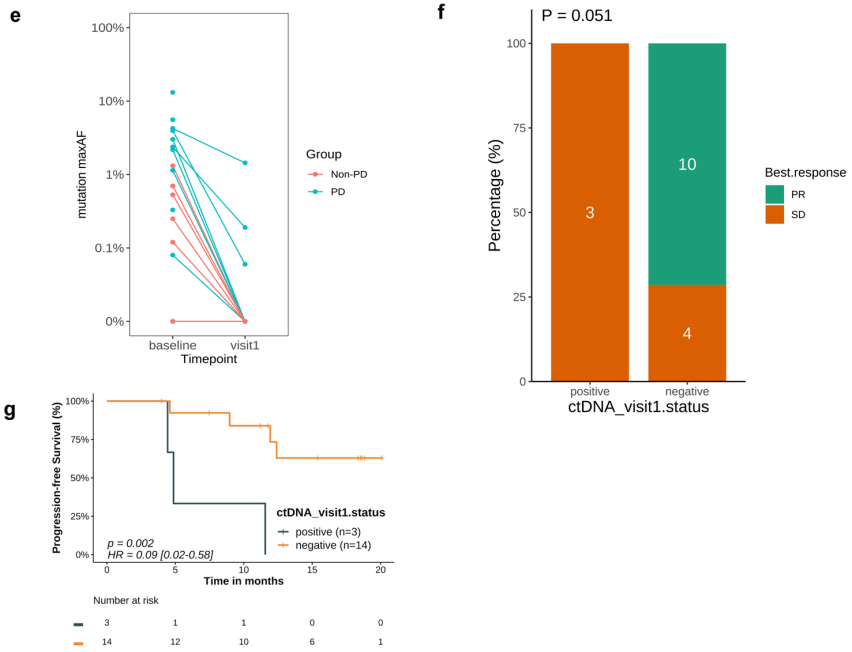

图1
治疗早期ctDNA动态变化可预测普拉替尼治疗效果:
所有患者在治疗第8周时均出现ctDNA水平下降(图1e)。治疗第8周ctDNA水平极低(maxAF<0.1%)或实现ctDNA清除(通过maxAF和MTM/mL两项指标检测,cfDNA中未发现肿瘤特异性变异)的患者,PFS显著延长,对应的P值分别<0.001和0.002(图1f)。且ctDNA清除与PFS的这种关联同样独立于治疗线数。ctDNA清除的患者ORR也更高(10/14例,缓解率71.4%;而未实现清除的3例患者缓解率为0%,见图1g),尽管这一差异未达到统计学显著水平,但呈现出相似的趋势。
三种不同的ctDNA动态模式与不同疾病进展结局相关:
除ctDNA清除外,其水平波动同样可反映肿瘤负荷的变化。三项ctDNA检测指标均与实体瘤疗效评价标准检测结果具有相关性。图2a汇总了患者关键时间点的临床结局与ctDNA检出情况。值得注意的是,ctDNA早期下降往往早于影像学上的疗效改善,这种情况在3例存在延迟但持久疗效的患者中尤为明显(见图2a、2b)。
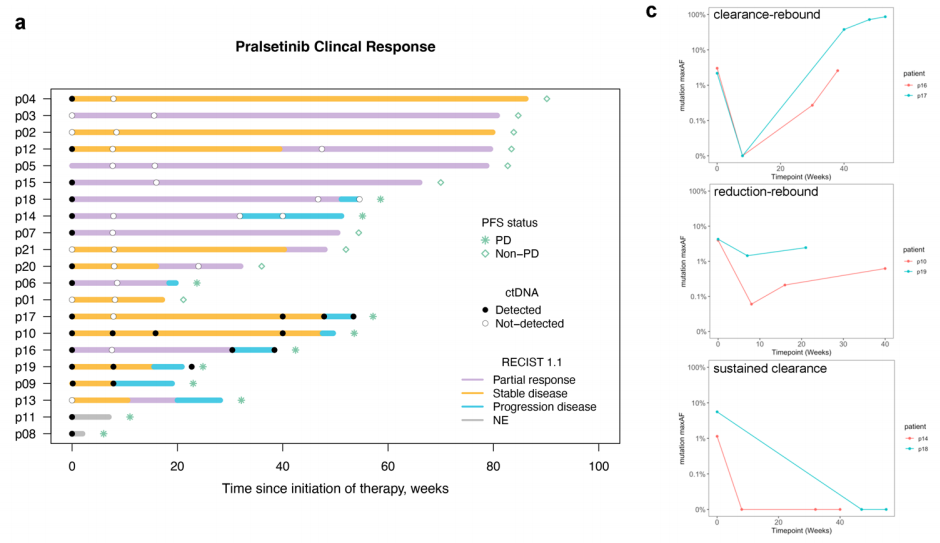
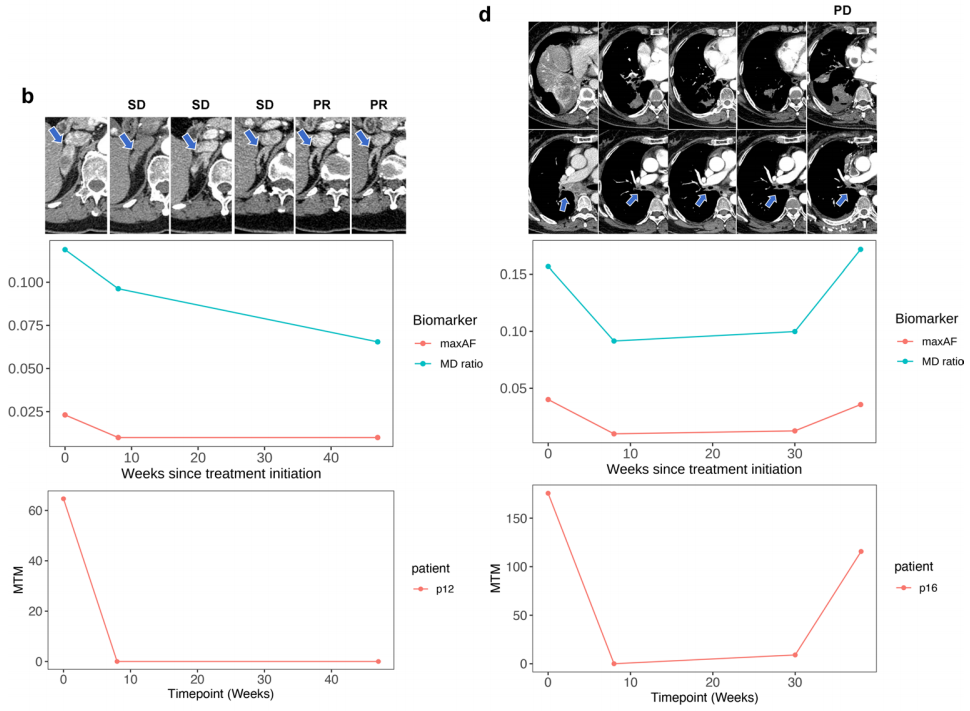
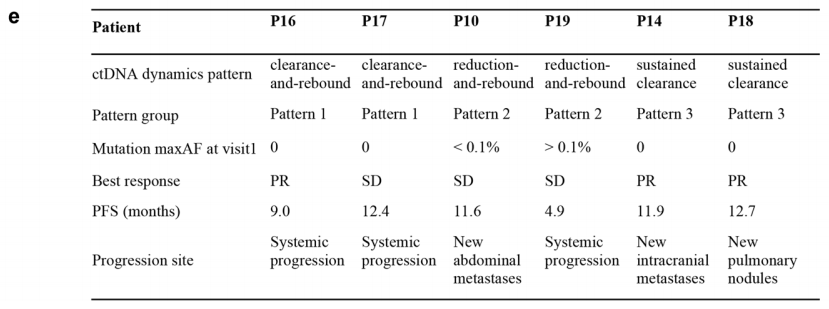

图2
研究中6例疾病进展患者的ctDNA呈现出三种不同的动态变化模式(图2c):一是清除 - 回升型,即治疗第8周ctDNA实现清除,但疾病进展时再次出现;二是下降 - 回升型,即治疗第8周ctDNA水平下降但仍可被检出,疾病进展时水平升高;三是持续清除型,即治疗第8周ctDNA实现清除,且疾病进展时仍未被检出。ctDNA回升的定义为,ctDNA经前期下降或清除后,其maxAF出现升高。
持续清除型患者的疾病进展部位多为ctDNA可能无法释放的区域,如双侧直径不足1厘米的肺结节或仅发生脑部病灶。这或许能解释为何此类患者体内未检测到ctDNA,但同时也不能排除其他可能性,例如肿瘤内或肿瘤间存在异质性、检测技术对极低频率突变的检出灵敏度有限,或是本研究检测panel未涵盖部分未被发现的分子变异等。参照既往针对EGFR突变NSCLC的研究标准,本研究对分子层面疾病进展的定义包含以下三种情况:其一,出现治疗前不存在的潜在耐药突变;其二,原有突变的等位基因频率较治疗前升高超过10%;其三,ctDNA清除后再次检测到任何变异(ctDNA清除指在当前检测技术的检出限下,未检测到ctDNA,或所有治疗前存在的突变均降至检测不到的水平,且该标准不限于RET融合突变)。分子层面的疾病进展较影像学确诊的疾病进展平均提前2.2个月(图2d)。
普拉替尼耐药后的基因突变特征:
耐药相关基因分析显示,患者耐药后出现KRAS G12R获得性突变,但未检测到RET靶点本身的变异。此外,还发现了其他获得性致癌变异,包括CSMD3基因扩增、B2M错义突变以及EPHA7 E239K突变。
本前瞻性研究整合等位基因频率、cfDNA定量标准化及甲基化相关三类ctDNA检测指标,对RET融合阳性NSCLC患者接受普拉替尼治疗后的疗效与疾病进展情况展开监测。研究证实,治疗前ctDNA水平较低以及治疗早期实现ctDNA清除的患者,PFS更长。这一结论与免疫治疗领域,以及针对EGFR、KRAS等靶点的靶向治疗相关研究结论一致。同时,相较于既往针对RET靶点的相关研究,本研究纳入了更多类型的ctDNA生物标志物,并详细描述了前瞻性队列中ctDNA的纵向动态变化特征,进一步完善了相关研究证据。本研究将新型检测指标与传统的等位基因频率检测相结合,证实这些指标与患者生存结局及肿瘤负荷均存在显著相关性。治疗前PIK3CA共突变对疗效产生的不良影响,既印证了该基因在肿瘤发生发展相关信号通路中的作用,也提示此类患者或可从联合治疗中获益,既往已有研究探索RET抑制剂与PI3K通路抑制剂联合用药的方案,也为此提供了依据。本研究中观察到的获得性耐药病例较少,这与既往相关研究结论相符。
但本研究仍存在一些局限性。首先,样本量较小(共21例),其中仅2例存在PIK3CA突变,这限制了亚组分析结果的可靠性,相关结论需在更大规模的独立队列研究中进一步验证。其次,尽管本研究明确了潜在的耐药机制,但未对这些机制开展功能验证。最后,上述三项ctDNA检测指标的预测价值,还需在接受同类治疗的外部独立队列中予以确认。综上,本研究结果为未来基于ctDNA指导的实时临床决策提供了参考。后续仍需开展更多临床试验,验证这些生物标志物的有效性,并将其纳入干预性研究设计中。
参考文献:
Lu C, Xu CR, Zhang YC, et al. Multiple ctDNA- based biomarkers predict benefit from selective RET Inhibition in non-small cell lung cancer patients: exploratory analysis of a prospective study. Biomark Res. 2025;13(1):98. Published 2025 Jul 23. doi:10.1186/s40364-025-00809-8
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)