首页 > 医疗资讯/ 正文
近年来,单细胞技术的高速发展显著推动了细胞研究,使单细胞转录组(scRNA-seq)、表观基因组(scATAC-seq)和蛋白质组等多维度分析成为可能。随着这些技术的不断突破和数据资源的积累,越来越需要能够整合不同模态信息的计算方法,以对单细胞多组学数据进行联合分析,从而更全面地了解细胞状态和功能。
目前,不同组学数据的整合面临跨模态特征关联性较弱、部分技术检测特征有限等挑战。现有整合计算方法多针对强关联组学特征或批次效应校正。因此,亟需开发兼顾生物信息保存与跨模态对齐的新框架。
为应对上述挑战,美国耶鲁大学研究团队开发了深度学习框架scMODAL,通过整合有限的特征关联(Feature Links)信息实现单细胞多组学数据的高效对齐,并保留特征拓扑结构。经不同多组学数据集验证,scMODAL在消除非必要变异、保留生物信息以及准确识别不同数据集细胞亚群方面效果显著,不仅推进了转录组-蛋白质组、转录组-染色质可及性等多组学整合任务,还支持特征插补和特征关系推断等下游分析,为单细胞多组学研究提供了全面且高效的工具。

scMODAL是一种基于特征关联的通用深度学习框架,可从单细胞多组学特征中学习整合的细胞表示,用于单细胞多组学数据对齐。其核心流程包括:
输入与特征配对:以细胞-特征矩阵为输入,利用先验知识将跨模态正相关特征(如基因表达与蛋白丰度)配对为S1/S2矩阵。
非线性特征映射:利用神经网络(编码器E1和E2)将不同模态数据(如scRNA-seq的X1和蛋白质组学的X2)映射至共享潜在空间z,保留全特征信息而非仅依赖共享特征。
生成对抗学习:采用生成对抗网络(GANs)最小化模态间细胞嵌入的分布差异(Jensen-Shannon散度),确保跨模态数据的混合均匀性。
锚点正则化:基于已知特征关联算互近邻(MNN)对作为锚点,通过L2惩罚项约束锚点嵌入距离,引导正确的细胞状态匹配。
几何结构保留:通过高斯核距离正则化,维持各模态内细胞间的相对距离,保留原始数据集的拓扑结构,避免过度拟合导致生物信息丢失。
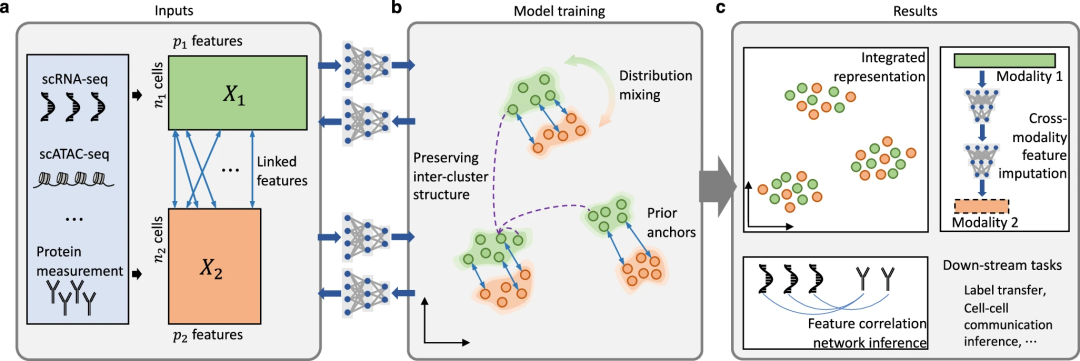
图1. scMODAL概述
研究团队首先在人类CITE-seq外周血单核细胞(PBMC)数据集中评估了scMODAL的性能,将其与MaxFuse、bindSC等多种整合方法比较,从细胞分布混合度、细胞类型分离度和细胞状态匹配准确性等方面进行分析。结果显示,scMODAL与其他跨模态整合方法的对齐性能相当,但在整合准确性方面表现最佳。
值得注意的是,scMODAL具有最高的标签转移准确率,分别为 98%(一级注释)、86%(二级注释);平均轮廓宽度(ASW)分数显著更高,具有保留自然杀伤细胞、CD4 T细胞等细粒度细胞群体的能力。
在弱关联模态的CITE-seq 数据(仅30个蛋白标记)中,scMODAL预测的蛋白质丰度与真实数据的平均相关性达0.53,显著优于其他方法。此外,在包含全转录组基因表达谱及97种Ab-seq表面蛋白标记的人类骨髓数据集中,scMODAL在整合转录组学与蛋白质组学等弱关联模态时实现了最高性能指标,凸显其优势。
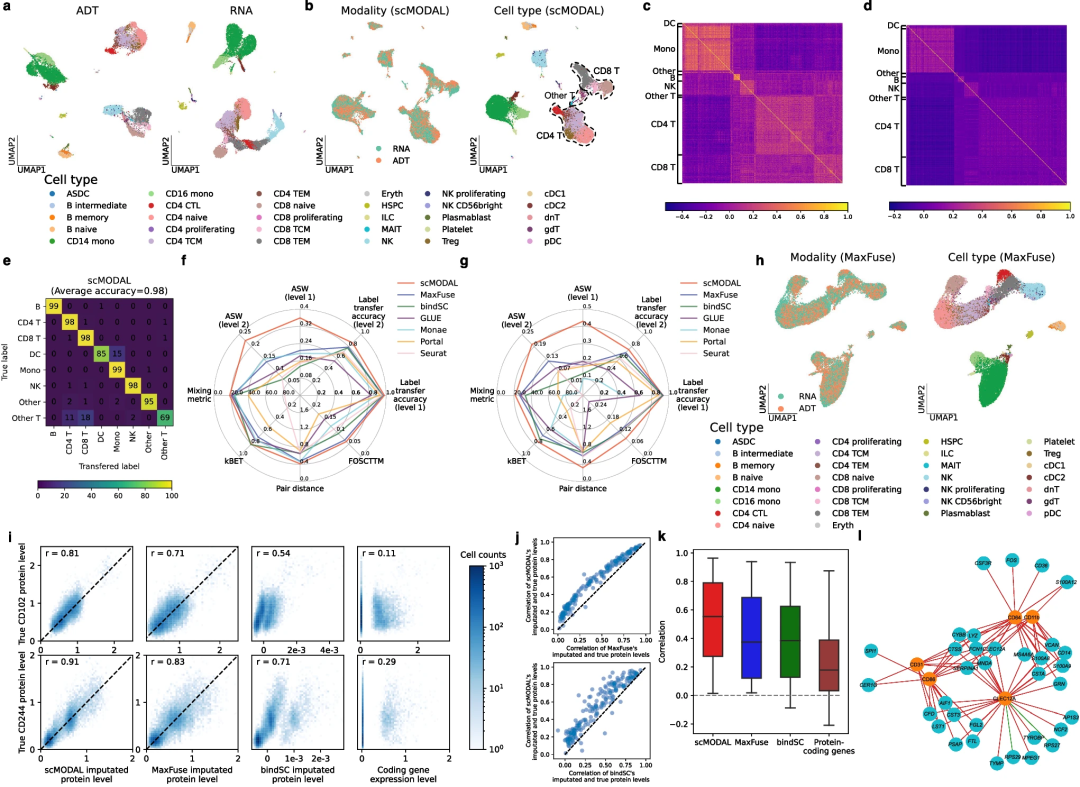
图2. 对CITE-seq产生的转录组和蛋白质数据进行整合的基准测试
为进一步验证scMODAL的有效性,研究团队在两种更具挑战性的场景中对其与其他整合方法进行了基准测试:1.共享特征极少的数据集;2.整合具有不同程度共享信息的多模态数据集。
在仅含12个共享标记的CITE-seq、CyTOF骨髓数据集中,scMODAL在整合嵌入空间中实现了优秀的混合效果,具有最高标签转移准确率;并产生了最佳的细胞类型分组(最高细胞类型轮廓系数),有效保留了生物学变异。
在来自同一人类PBMC样本、包含转录组、蛋白组和染色质可及性(TEA-seq)的三模态数据中(TEA-seq PBMC),scMODAL成功保留B细胞、T细胞等簇结构,具有较高的跨模态标签转移准确率,RNA到ADT/ATAC的标签转移准确率分别达87%和83%,凸显了scMODAL处理复杂异质性跨模态整合任务的强大能力。

图3. scMODAL在有限共享特征及三模态数据集中的应用
研究团队还在复杂组织(脑、扁桃体)的多模态数据中评估了scMODAL的跨模态整合性能。
首先利用scMODAL整合了从小鼠大脑皮层获得的scRNA-seq数据集和scATAC-seq数据。结果显示,相同类型细胞在整合空间中正确对齐;Louvain聚类识别出15个细胞亚群(包括9种神经元亚型),跨模态相同簇的细胞具有高相关性;皮层层特异性基因(如Lamp5、Rorb)表达模式在跨模态数据中高度一致,表明scMODAL成功保留了皮层神经元亚群的精细结构。
通过基因表达插补,scMODAL还纠正了scATAC-seq基因活性预测的偏差(如Fam107a在星形胶质细胞中的特异性表达),并推断出其潜在调控峰,证明了该方法在解析复杂器官调控网络中的价值。
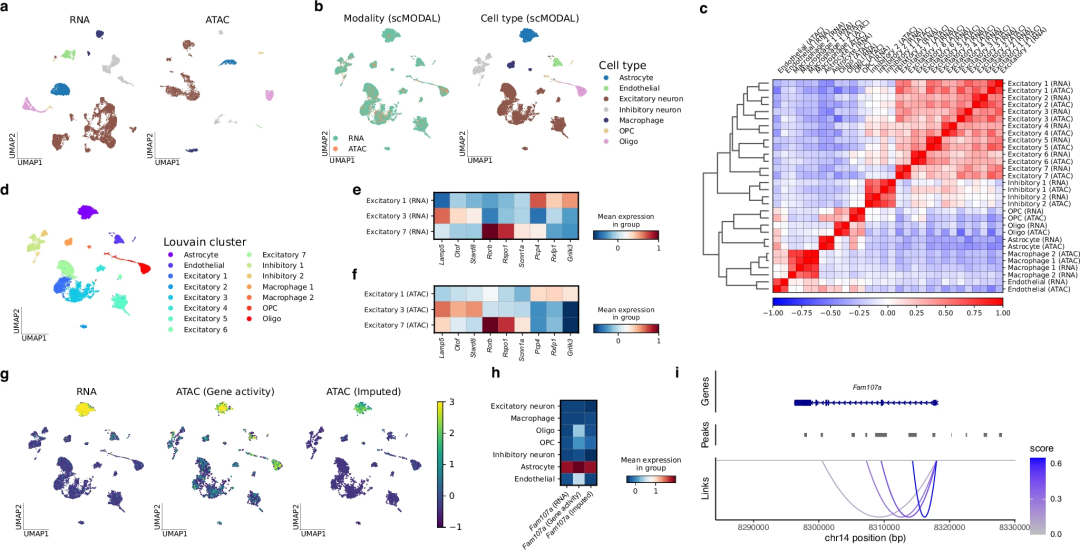
图4. 整合小鼠大脑scRNA-seq和scATAC-seq数据集
在人扁桃体CODEX空间蛋白组、scRNA-seq和scATAC-seq多模态数据整合研究中,scMODAL成功将scRNA-seq标注的细胞类型标签(如生发中心B细胞、边缘区B细胞)转移至CODEX空间蛋白组和 scATAC-seq数据中,揭示了B细胞滤泡的空间结构:B-Ki67细胞聚集形成生发中心外围,由B-CD22-CD40细胞构成边缘区环状结构。
scMODAL通过插补MKI67基因表达,准确捕捉到在生发中心高表达并向外递减的B细胞增殖梯度空间特异性模式;利用插补的CODEX基因表达,推断出CCL4-SLC7A1等细胞间通讯通路,展示了其在空间多组学分析和免疫微环境机制研究中的应用潜力。

图5. 整合人扁桃体CODEX、scRNA-seq和scATAC-seq数据集
综上所述,scMODAL通过深度学习突破了单细胞多组学整合的技术瓶颈,有效解决了跨模态整合的关键挑战,为研究人员探索不同细胞成分之间的复杂相互作用提供了强大工具。
参考文献:
Wang, G., Zhao, J., Lin, Y. et al. scMODAL: a general deep learning framework for comprehensive single-cell multi-omics data alignment with feature links. Nat Commun 16, 4994 (2025). https://doi.org/10.1038/s41467-025-60333-z
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)