首页 > 医疗资讯/ 正文
导读
肝细胞癌(hepatocellular carcinoma, HCC),特别是原发性 HCC,是全球癌症相关死因死亡的主要原因之一。临床上,对伴有其他癌症病史的患者,鉴别肝脏肿瘤是原发性肝癌还是转移性病灶,如结直肠癌肝转移(metastatic colorectal cancer, mCRC),往往具有挑战性。虽然传统的影像学检查、血清学标志物如甲胎蛋白(alpha- fetoprotein, AFP)以及组织活检是常规诊断方法,但仍存在一定局限性。
近日,美国加州大学圣地亚哥分校Amir Zarrinpar教授团队在eGastroenterology上发表了题为“Blood microbial DNA signature differentiates hepatocellular carcinoma from metastatic lesions”的短篇报道,通过分析血液微生物游离DNA(cell-free DNA, cfDNA)特征,开发出一种“微生物指纹”检测方法,能够有效区分HCC与mCRC患者,为临床诊断提供了新的无创分子检测策略。

微生物与癌症:一个日益受到关注的新视角
近年来,肿瘤微生态(onco-microbiome)研究进展迅速。多项研究表明,肿瘤组织中存在与宿主基因表达相互作用的细菌和真菌等微生物,它们不仅可能参与肿瘤的发生发展,还可能影响免疫治疗和化疗的效果。对癌症基因组图谱(The Cancer Genome Atlas, TCGA)中血液和组织样本的全基因组测序数据分析发现,微生物群落特征能够区分HCC与非癌组织及其他癌症类型。循环微生物组DNA在肺癌的早期诊断中也展现出巨大潜力。然而,目前尚未有研究能够明确微生物cfDNA是否可用于区分原发性肝癌与转移性肝癌。
研究设计:聚焦微生物cfDNA的分型能力
本研究纳入16例HCC患者与11例mCRC患者。所有患者均为非活动性乙肝病毒感染状态,以排除既往研究中乙肝病毒DNA可能造成的干扰。
研究团队采用高通量“鸟枪法”宏基因组测序技术,对患者的肿瘤组织、癌旁组织和血浆样本中的微生物序列进行全面检测,并通过多种预处理进行去污染和去宿主序列等。关键步骤包括:使用Human Pangenome Reference过滤宿主DNA、采用QIITA平台进行微生物分类以及使用SCRub去除实验室污染物(见图1)。
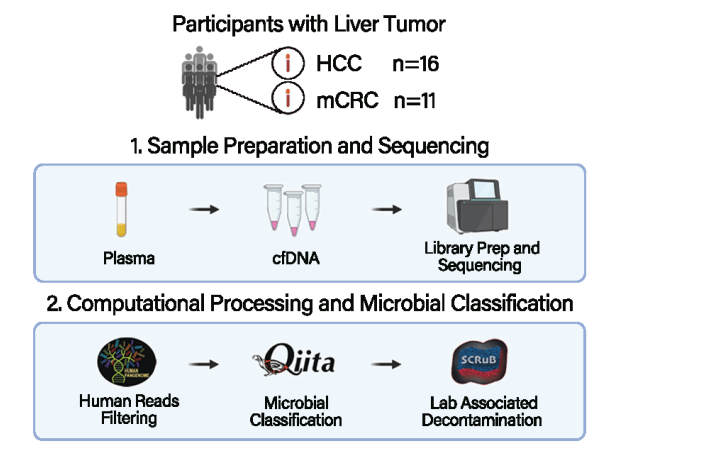
图1:该图展示了对肝脏肿瘤患者血浆cfDNA中微生物特征分析流程。研究共纳入27例患者,包括16例HCC和11例mCRC患者。分析流程包括:1)血浆cfDNA提取、文库构建以及高通量测序;2)去除宿主序列、采用Qiita平台进行微生物分类以及使用SCRuB工具去除实验室污染,最终获得可信度较高的血浆cfDNA微生物组成数据
关键发现:血浆微生物cfDNA特征可清晰区分HCC与mCRC
稳健主坐标分析 (Robust Principal Coordinates Analysis , RPCA) 和置换多元方差分析 (Permutational Multivariate Analysis of Variance, PERMANOVA) 结果显示,HCC与mCRC患者的血浆微生物组成存在显著差异,这一结果可能是因为mCRC患者(同时携带结直肠和肝脏肿瘤微生物)比HCC患者(仅携带肝脏肿瘤微生物)具有更强的血浆微生物信号。然而,肿瘤组织与癌旁组织的微生物组成在两组间未发现显著差异,这提示:血液微生物信号可能更敏感、更具诊断潜力;组织微生物受局部环境、低生物量和技术干扰的影响更大,可能难以捕捉差异(见图2)。通过贝叶斯推断回归微生物组差异分析 (Bayesian Inference of Relative Differences in Microbiome Analysis, BIRDMAn) 方法,研究团队进一步鉴定出两类肿瘤的微生物“指纹”:
-
mCRC富集菌群:包括不动杆菌属 (Acinetobacter, 如A. septicus、A. parvus)、亚洲假单胞菌 (Pseudomonas asiatica) 和粪双歧杆菌(Bifidobacterium faecale)。其中,不动杆菌属和亚洲假单胞菌与院内感染相关,而粪双歧杆菌在动物模型中显示与抗肿瘤免疫相关。
-
HCC富集菌群:包括铜绿假单胞菌 (Pseudomonas aeruginosa) 及棒状杆菌属 (Corynebacterium, 如C. accolens、C. glucuronolyticum),这些菌株不仅与免疫抑制状态和肝移植并发症相关,还可能通过抑癌通路或抗菌防御机制影响肝脏微环境。
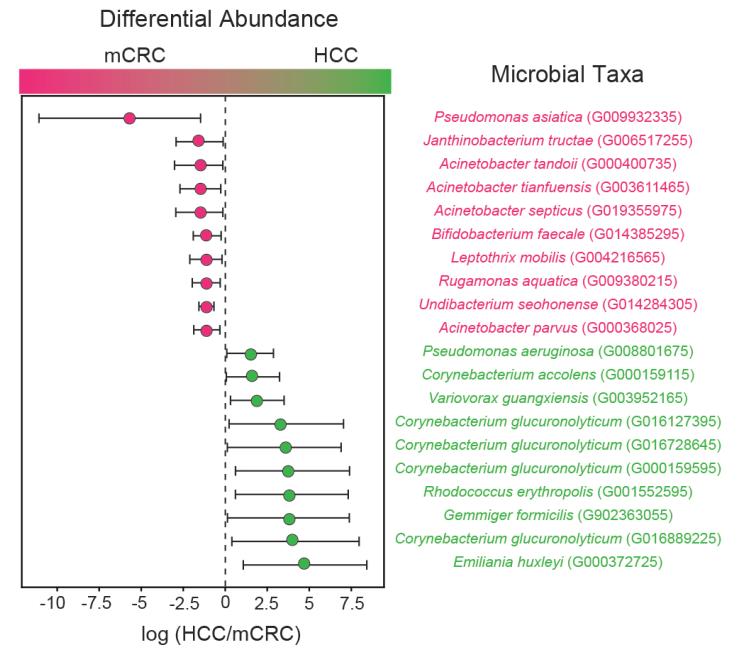
图2:该图展示HCC与mCRC患者血浆中微生物丰度的差异分析结果。横轴为微生物在HCC与mCRC之间的对数丰度比(log(HCC/mCRC)),越向左代表在mCRC中越富集(粉红色),越向右代表在HCC中越富集(绿色)。图中展示了若干具有统计学显著差异的微生物分类单元。这些微生物组差异可能反映不同肿瘤类型的特异微生物组特征,为进一步研究肿瘤-微生物相互作用提供线索
机制推测:血液微生物cfDNA为何能揭示肿瘤类型?
血液微生物cfDNA的来源可能包括肝脏、肠道或口腔等。在mCRC患者中,CRC及其转移过程可能导致肠道屏障破坏,使细菌经门静脉系统进入肝脏,释放cfDNA进入循环,因此在血液中会形成更强的微生物信号。而HCC患者多伴有肝硬化背景,其微生态特征可能不同于无肝脏疾病的mCRC患者。尽管组织样本中未观察到显著的微生物组差异,但血浆微生物组差异表明其在区分肿瘤类型方面具有潜力。
临床启示与未来方向:迈向精准化、无创肿瘤诊断
本研究具有重要临床价值。相比传统的肿瘤标志物,如AFP,存在灵敏度局限性(本研究中仅1例HCC患者AFP > 400 ng/mL),而血浆微生物cfDNA提供了一个全新的无创检测维度,特别适用于AFP阴性或影像学诊断不明确的病例。
未来的潜在应用包括:
-
HCC与mCRC的鉴别诊断:尤其适用于影像表现不典型或无法进行活检的患者。
-
早期肿瘤筛查:通过扩大样本量并优化测序深度,有望发展为早筛工具。
-
疗效监测与预后评估:随访血液微生态变化,可能反映出肿瘤负荷的消长或治疗响应的优劣。
-
微生态干预治疗:结合微生物靶点,未来或可发展“调菌治癌”的策略。
未来需要在高危人群、早期患者和不同肝脏疾病背景的人群中,开展多中心、大样本、前瞻性的研究。同时,还需结合口腔、肠道以及肝脏组织等多部位的微生态图谱,全面理解其生物学意义与诊断价值。
结语
微生物cfDNA已成为肿瘤诊断领域的新兴方向。本研究不仅为HCC与mCRC的鉴别诊断提供了潜在工具,也为发展“血液微生物指纹”在肿瘤早筛、风险预测和个体化干预中的应用奠定了基础。随着测序成本降低和算法优化,这一技术有望从实验室走向临床,为肝癌等高致死性肿瘤的精准诊疗开启新篇章。
引证本文
Guccione C, Dantas Machado AC, Youssef F, et al. Blood microbial DNA signature differentiates hepatocellular carcinoma from metastatic lesions. eGastroenterology 2025;3:e100193. doi:10.1136/egastro-2025-100193
https://doi.org/10.1136/egastro-2025-100193
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)