首页 > 医疗资讯/ 正文
碳青霉烯耐药肺炎克雷伯菌(CRKP)是极具威胁性的医院获得性感染病原体[1-2],具有高度的环境适应性和传播能力,常通过质粒、转座子等可移动遗传元件实现耐药基因的横向转移,从而促进耐药菌株的繁殖与扩散,加速耐药性在菌群间的传播[3]。既往研究显示[4],胃肠道无症状定植的CRKP是继发侵袭性感染的重要独立危险因素。
无症状定植患者在机体免疫低下或接受侵入性操作时,极易进展为严重的院内感染,其中血流感染发生率较非定植患者增高2倍[5]。目前,虽然CRKP流行病学特征已有诸多报道,但关于定植菌株和感染菌株之间的遗传关系及其动态变化研究仍相对薄弱。基因组学不仅为揭示二者之间的遗传关联提供了高分辨率工具,也是深入阐明其流行趋势与医院内传播机制的关键手段[6-8]。本研究通过结合临床资料、耐药表型、毒力特性及基因组学分析,系统阐明CRKP肠道定植与血流感染菌株之间的关联性,从而为CRKP去定植方案及抗感染治疗策略的制定提供科学依据。
1 材料与方法
1.1 一般材料
本研究为回顾性分析,收集2023年1月—2024年12月昆明医科大学第一附属医院碳青霉烯耐药肠杆菌目细菌(CRE)筛选阳性住院患者的临床资料。分析其直肠拭子样本及住院期间感染情况。对于发生血流感染的患者,采集其血液标本并分离血流感染菌株,将同一患者的肠道定植菌株与其血流感染菌株进行配对分析。
纳入标准:(1)严格按照美国疾病控制与预防中心(CDC)医院感染防控指南[9]进行CRE主动筛查;(2)所有菌株经基质辅助激光解吸电离飞行时间质谱仪(MALDI-TOF MS)鉴定为肺炎克雷伯菌;(3)感染菌株符合《医院感染诊断标准》[10]中的诊断标准。
排除标准:(1)肠道定植菌与血流感染菌菌种不一致;(2)同一患者在相同部位的重复分离菌株。
本研究已通过昆明医科大学第一附属医院医学伦理委员会批准[伦理号:(2024)伦审L第257号]。
1.2 抗菌药物敏感性试验
按照2023年美国临床和实验室标准化协会(CLSI)推荐的药敏试验要求,使用VITEK Ⅱ全自动微生物鉴定及药敏分析仪对肠道定植菌株与血流感染菌株进行抗菌药物敏感性试验。针对替加环素、多黏菌素B及头孢他啶/阿维巴坦,采用微量肉汤稀释法测定最低抑菌浓度(MIC)。实验结果参照CLSI M100-S33文件[11]推荐的标准进行判读。多黏菌素B药敏结果按照欧洲抗菌药物敏感性测试委员会标准(http://www.eucast.org)进行判读。替加环素药敏结果采用美国食品药品监督管理局判读标准[12]。质控菌株大肠埃希菌ATCC25922由国家卫生健康委员会临床检验中心提供。
1.3 结晶紫染色法半定量生物被膜形成能力
所有实验菌株经纯培养后,取单个典型菌落,制备成0.5麦氏浓度(相当于1.5×108 CFU/mL)菌悬液,并以Luria-Bertani(LB)液体培养基为阴性对照,在37 ℃条件下静置培养24~48 h。弃上清液,依次进行PBS缓冲液洗涤3次,甲醇溶液固定15 min,1% 结晶紫溶液染色15 min,蒸馏水洗涤3次,95%乙醇脱色15 min。采用酶标仪在595 nm波长处测定各孔光密度(OD)值。根据OD595测定结果,判定并评价菌株的生物被膜形成能力。
1.4 铁载体定性定量试验
所有菌株经饥饿处理后,取单个菌落,用蛋白胨水配制成4麦氏浓度的菌悬液,并取500 μL加至含2.5 mL液体蛋白胨培养基中。混匀后于37 ℃,220 r/min恒温摇床中培养24~48 h。以蛋白胨为空白对照,测定600 nm波长处OD值(OD600)评估菌株生长情况。剩余菌液予以5000 r/min离心15 min处理,并过滤上清液。将100 μL滤过上清和100 μL预配置的铬天青S(CAS)定量检测液混匀,于37 ℃孵育1 h,然后采用酶标仪测定OD630,根据吸光度变化计算铁载体产量。
1.5 血清杀伤试验
收集40名健康体检志愿者的血清,每名志愿者留取500 μL,混合后备用。将所有待测菌株制备成0.5麦氏浓度菌悬液,并采用PBS缓冲液稀释100倍。取500 μL稀释的菌液接种至含有500 μL 混合人血清的EP管中,于37 ℃ 200 r/min的振荡条件下培养,并在0 h、1 h、2 h和3 h分别进行活菌计数。通过比较各时间点的菌落数,判断菌株的抗血清能力。
1.6 全基因组测序
将直肠拭子阳性菌株和继发血流感染的CRKP菌株送至上海美吉生物医药科技有限公司进行全基因组(WGS)测序,方法为二代测序(Illumina NovaSeqTM Ⅹ Plus平台)和三代测序(PacBio Sequel IIe平台。
为确保后续分析的准确性,使用FastQC vO.11.2软件对测序数据质量进行评估,去除测序过程中产生的接头序列、低质量“reads”及含有过多“N”的“reads”,然后使用fastx_toollkit软件包进行过滤处理,最终获得高质量“clean reads”。
利用SPAdes软件基于denovo基因组装原理对“clean reads”进行基因组组装,以获得完整的基因组序列,然后通过RAST平台进行基因预测和功能注释分析;同时,结合BLAST工具对基因序列进行比对分析。
1.7 生物信息学分析
将所有实验菌株全基因组序列上传至基因组流行病学中心(CGE)平台(https://genomicepidemiology.org/)进行如下生物信息学分析:
使用ResFinder工具鉴定菌株携带的耐药基因,设置相似性阈值为90%,覆盖度为60%;
使用VFDB工具进行肺炎克雷伯菌相关毒力基因的识别;
通过MLST 2.0在线工具确定菌株的序列型(ST);
使用PlasmidFinder工具识别所有质粒复制子类型。全质粒序列由Prokka 1.14.6软件注释,并借助BRIG软件进行质粒圈图可视化;
使用Kleborate 2.0.0在线工具[13]对菌株进行K型及O型抗原分型,准确确定其血清型。
1.8 比较基因组学分析
1.8.1 基于核心基因组单核苷酸多态性热图与系统发育树分析
以最早收集的患者肠道定植菌株(CRKP4-re)作为参考菌株,使用Snippy软件与其余实验菌株全基因组测序数据进行比对,筛选高质量核心基因组单核苷酸多态性(SNP)位点。SNP矩阵经Gubbins去同源重组处理后,利用IQ-TREE软件基于最大似然法(ML)构建系统发育树,并通过1000次自助法(bootstrap)重采样评估各分支的置信度,从而探讨院内菌株的传播与进化过程。在此基础上,将核心SNP距离矩阵导入R语言软件,绘制菌株间遗传差异热图。
1.8.2 基于核心基因组多位点序列分型的最小生成树分析
通过核心基因组多位点序列分型(cgMLST)数据库,提取cgMLST等位基因型数据。使用GrapeTree软件,根据菌株间等位基因数目差异构建最小生成树(MST)。
1.9 统计学处理
采用SPSS 27.0软件进行统计学分析。使用Cohen's Kappa计算肠道定植菌株和血流感染菌株耐药谱的分类一致性(CA),以评估来自同一患者的菌株耐药性一致性;同时,计算组内相关系数(ICCs),以分析二者毒力表型的一致性。
2 结果
2.1 菌株检出情况及患者基本信息
研究期间共12 878例患者进行CRE筛查,共发现肠道定植CRKP 60例,其中6例出现血流感染,即CRKP肠道定植继发血流感染发生率为10%。发生血流感染的患者中,来自重症监护病房4例(男性3例,女性1例)、血液内科1例(女性)、老年呼吸内科1例(男性),年龄范围为42~93岁,平均年龄70.5岁。6例患者均接受了抗菌治疗,以及机械通气、泌尿道置管等侵入性医疗操作,住院期间死亡4例(66.7%)。6例CRKP肠道定植继发血流感染患者的基本信息见图1。
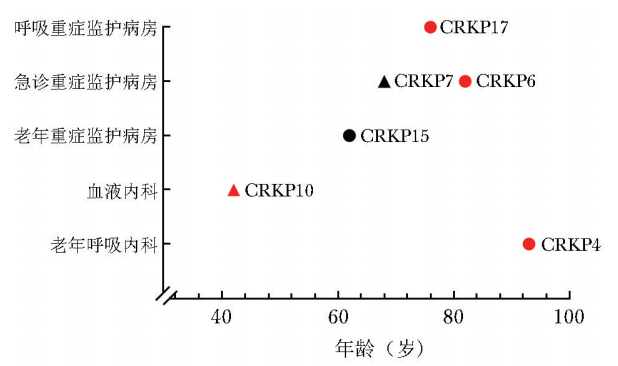
图1 CRKP肠道定植继发血流感染患者基本信息
颜色反映预后结局(红色:死亡;黑色:好转),形状区分性别(▲:女性;●:男性),菌株末尾数字表示患者编号
CRKP :碳青霉烯耐药肺炎克雷伯菌
2.2 菌株耐药性与毒力表型分析
通过分析6对肠道定植菌株与配对血流感染菌株的抗菌药物耐药性,除来自患者7的菌株(CA=82.6%)外,余肠道定植菌株与血流感染菌株在除替加环素之外的其他抗菌药物的耐药谱上表现出高度一致性,CA均达100%(图2)。
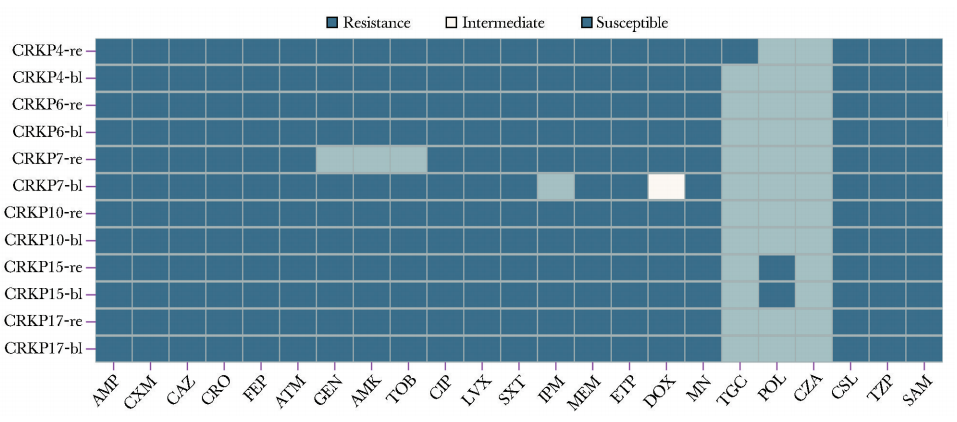
图2 6对肠道定植菌株和血流感染菌株药敏结果一致性分析
菌株编号末位数字相同表示样本来源于同一患者,其中“re”代表其肠道定植菌株,“bl”血流感染菌株;AMP:氨苄西林;CXM:头孢呋辛;CAZ:头孢他啶;CRO:头孢曲松;FEP:头孢吡肟;ATM:氨曲南;GEN:庆大霉素;AMK:阿米卡星;TOB:妥布霉素;CIP:环丙沙星;LVX:左氧氟沙星;SXT:复方新诺明;IPM:亚胺培南;MEM:美罗培南;ETP:厄他培南;DOX:多西环素;MN:米诺环素;TGC:替加环素;POL:多黏菌素B;CZA:头孢他啶/阿维巴坦;CSL:头孢哌酮/舒巴坦;TZP:哌拉西林/他唑巴坦;SAM:氨苄西林/舒巴坦;CRKP:同图1
这些菌株普遍对替加环素、多黏菌素B及头孢他啶/阿维巴坦敏感,但也有个别耐药现象,例如患者4的肠道定植菌株(CRKP4-re)对替加环素耐药,患者15的肠道定植菌株(CRKP15-re)及其配对感染菌株(CRKP15-bl)对多黏菌素B耐药。除耐药谱一致性外,配对的肠道定植菌株与血流感染菌株在毒力表型方面也表现出高度一致性。二者的生物被膜形成能力和铁载体产量的ICCs分别为0.8770~0.9768和0.7905~0.9921,其数值均高于0.75,提示具有明显的毒力表型一致性(图3A、3B)。所有菌株均具有强阳性生物被膜形成能力及较高的铁载体产量(>30%)。在免疫逃逸能力方面,所有菌株在人体正常血清中的生存能力均较弱,培养3 h后被完全清除(图3C)。
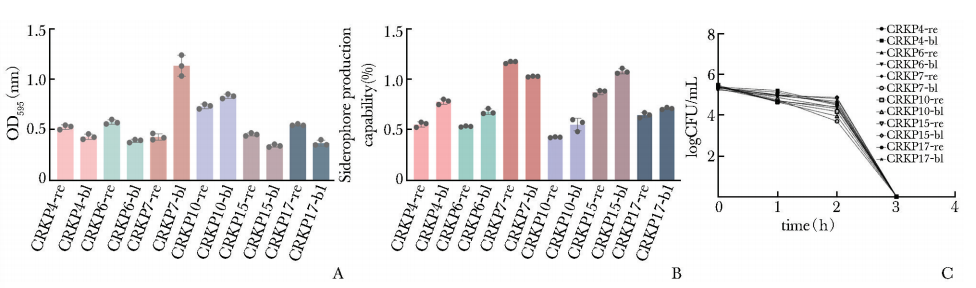
图3 定植菌株与血流感染菌株毒力表型的一致性分析
A.肠道定植菌株与血流感染菌株生物被膜形成能力比较;B.肠道定植菌株与血流感染菌株铁载体生成能力比较;C.12株CRKP血清杀伤能力检测结果
CRKP:同图1
2.3 全基因组测序和比较基因组学分析
2.3.1 基因特征
WGS分析发现,所有菌株中以ST11-KL64型产KPC-2酶为主要类型(91.7%,11/12),仅患者7的血流感染菌株(CRKP7-bl)为ST231-KL51型产OXA-232酶的CRKP。值得注意的是,患者15的血流感染菌株(CRKP15-bl)中blaKPC-2基因在感染过程中整合至染色体上(图4A),而其余菌株的碳青霉烯酶耐药基因均位于IncFⅡ型质粒(图4B)。
所有菌株均检测到超广谱β-内酰胺酶(ESBLs)基因及多类耐药基因,表现出广泛的多重耐药性(图4C)。
在毒力基因方面,IncHI1B型毒力质粒携带的6个毒力基因(rmpA、rmpA2、iucA、iutA、iroB、peg344)在肠道定植CRKP与配对血流感染菌株中高度保守。其中,iutA和iucA携带率最高(91.7%,11/12),rmpA2次之(83.3%,10/12),而rmpA和peg344携带率均为50%(图4D)。此外,在染色体上检测到多种铁载体相关基因(如fep、ybt)和菌毛相关基因(如mrk、fim),这些基因的存在可能与菌株致病能力密切相关(图4E)。
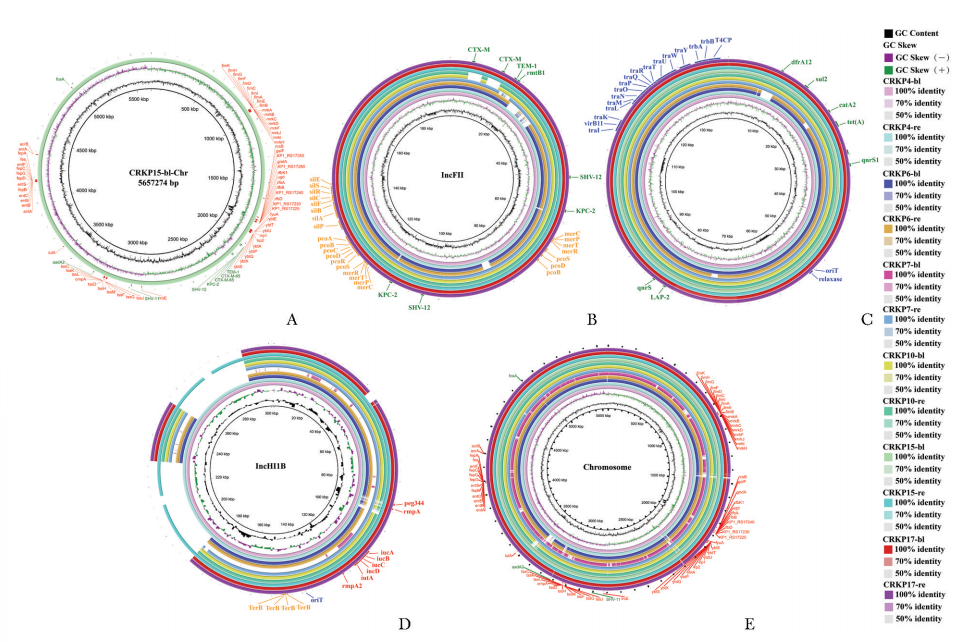
图4 质粒和染色体结构图
A.患者15感染菌株(CRKP15-bl)染色体圈图;B.IncFⅡ型耐药质粒圈图;C.其他耐药质粒圈图;D.IncHI1B型毒力质粒圈图;E.所有菌株染色体圈图;黄色为金属抗性基因,红色为毒力基因,绿色为耐药基因,蓝色为接合系统相关基因;不同颜色的圈环对应不同的菌株
CRKP:同图1
2.3.2 系统发育分析
基于核心基因组SNP构建的系统发育树显示,除患者7外,同一患者的肠道定植菌株与血流感染菌株在进化树中均位于同一分枝,且分支长度<1×103,SNP差异小于10,表明二者具有高度的基因同源性。此外,不同科室来源患者的肠道定植菌株亦显示一定的克隆相关性(SNP<120)(图5A、5B)。
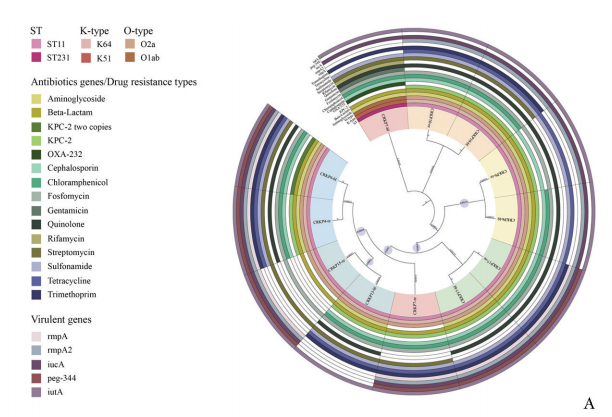
图5A 基于核心基因组SNP构建的系统发育树,每个菌株分支上的数字表示该菌株相较于其最近共同祖先的进化变异的程度,数字越小表示差异越小、进化距离越近,蓝色圆圈大小代表每个节点的自助法(bootstrap)支持度置信度,用于指示分支距离的可信度及进化关系的可靠性,圆圈越大表示可信度越大;外圈显示各菌株所携带的基因特征
SNP:单核苷酸多态性;cgMLST:核心基因组多位点序列分型;CRKP:同图1
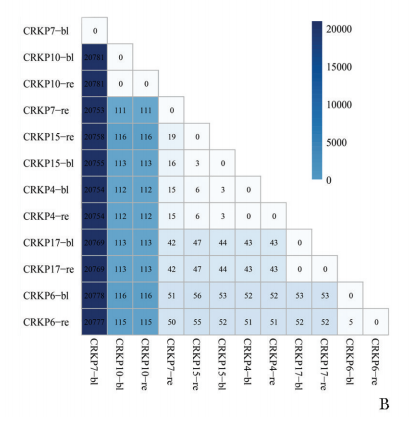
图5B 基于核心基因组SNP的热图,颜色深浅表示不同样本间核心基因组SNP位点的差异程度。颜色越深,即遗传差异越大;颜色越浅,表示SNP数量越少,遗传距离较近
SNP:单核苷酸多态性;cgMLST:核心基因组多位点序列分型;CRKP:同图1
基于cgMLST的MST分析显示(图5C),同一患者的肠道定植菌株与血流感染菌株聚类紧密,其中患者10和患者15的肠道定植菌株与血流感染菌株核心基因组之间未发现遗传差异位点。
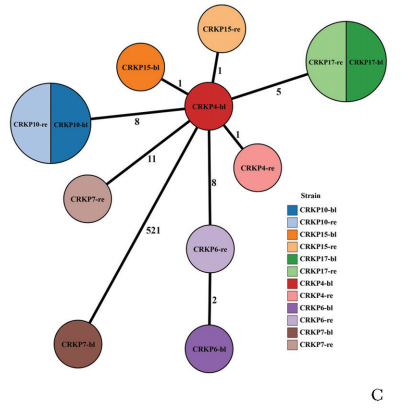
图5C 基于cgMLST构建的最小生成树,分枝长度取对数(log)进行处理,分支上的数字表示各菌株间的基因差异数量,分支长度反映亲缘关系距离(分支越短表示菌株亲缘关系越近,分支越长则表明亲缘关系越远),不同颜色区分不同菌株,基因无差异(即差异个数为0)的菌株在图中合并显示单个圆圈,如CRKP10-re与CRKP10-bl
SNP:单核苷酸多态性;cgMLST:核心基因组多位点序列分型;CRKP:同图1
部分患者的菌株样本之间形成独立分支,提示这些患者可能感染了遗传背景不同的菌株,但仍具有相对较近的克隆亲缘性。特别是患者7血流感染菌株(CRKP7-bl)的遗传距离最远(达521),显示其遗传背景显著不同于其他菌株。
3 讨论
本研究系统收集CRKP肠道定植及其配对的血流感染菌株,从耐药基因谱、毒力因子、可移动遗传元件及系统进化等角度,解析了CRKP遗传同源性与分子特征。结果发现,肠道定植菌株与血流感染菌株在基因组水平高度同源,耐药和毒力谱基本一致,基因型和表型均证实肠道定植CRKP是血流感染的直接来源。院内ST11-KL64型产KPC-2酶CRKP高度流行,且存在跨科室克隆传播风险。部分菌株表现出耐药基因染色体整合现象,反映出CRKP定植在感染进化中的动态多样性。
CRKP导致的血流感染因增长速度快、患者死亡率高,已成为全球范围内重大公共卫生挑战[14]。全国血流感染细菌耐药监测数据显示,我国血流感染病原菌中肺炎克雷伯菌检出率由2019年的17%迅速上升至2022年的25.9%[15],而且CRKP引发的血流感染死亡率高达40%~60%[16-17]。既往研究认为,肠道定植CRKP可能是其血流感染的诱发因素[4],但尚缺乏详实证据,尤其来自基因方面的关联性分析。
本研究系统性追踪了2023—2024年昆明医科大学第一附属医院12 878例进行主动筛查CRE患者的肠拭子样本数据,发现60例(0.47%)存在肠道定植CRKP,经持续监测其中6例(10.0%)继发血流感染,此类患者住院期间死亡率为66.7%,提示虽然CRKP肠道定植阳性率较低,但存在一定的血流感染风险且感染后死亡率高。
CRKP肠道定植血流感染主要集中于高危科室,包括重症监护病房、血液内科及老年病房。提示应需加强对此类高危科室的感控管理,以降低因CRKP定植而继发血流感染的风险。早期合理应用抗感染药物可显著改善CRKP血流感染患者的预后[18-19]。本研究发现,肠道定植CRKP菌株的药敏结果与血流感染CRKP菌株的药敏结果高度一致,提示当肠道定植CRKP引发血流感染时,可依据肠道定植的药敏结果,尽早调整抗生素用药方案,以实现精准治疗,改善患者临床结局。
本研究通过全基因组测序方法,从分子视角揭示CRKP的流行病学特征。结果显示,ST11-KL64型CRKP菌株在本中心肠道定植及院内血流感染中占据绝对优势,这与亚洲地区的流行趋势一致[20-21]。所有CRKP菌株均携带碳青霉烯酶基因(blaKPC/blaOXA-48)及超广谱β-内酰胺酶基因(blaCTX-M、blaSHV、blaTEM),同时携带了多种其他抗生素耐药基因,如氨基糖苷类(rmtB、aadA2)、喹诺酮类(oqxA/B、qnrS)、磺胺类(sul)等,呈现复杂多耐药基因谱,极大增加了临床治疗难度。与此同时,菌株还广泛携带铁载体相关基因(iutA、iucA、fep、ybt)和菌毛相关基因(mrk、fim),增强了其在铁限制环境下的定植、黏附及免疫逃避能力,有助于在肠道内持续存活并占据主导地位。
ST11-KL64型CRKP通过携带多种毒力基因与耐药基因整合,在肠道定植和后续感染过程中表现出较强的致病性、耐药性和适应性,显著提升定植继发血流感染的风险,给临床防控和管理带来严峻挑战。
系统发育分析显示,同一患者肠道定植菌株与血流感染菌株具有高度遗传同源性(SNP差异<10),且部分病例的两组菌株中未检测到遗传差异,明确证实了自体肠道定植CRKP菌株是侵袭性感染的重要直接来源。此外,肠道定植菌株与血流感染菌株在耐药和毒力表型上高度一致,进一步支持了上述结论,这与Henoun Loukili和Hu等学者的研究结论相符[22-23]。同时,不同科室来源患者的菌株之间存在克隆相关性(SNP差异<120),提示院内存在区域性传播,这可能与医护人员接触、医疗设备或环境污染相关,从而增加了交叉感染的风险[24]。因此,分区防控、集中消杀及多渠道切断传播链对于遏制高危克隆株的流行尤为必要。
值得注意的是,本研究发现菌株在从定植到感染过程中出现耐药与毒力质粒丢失的情况,而且相关基因可整合至染色体,反映出CRKP菌株在适应宿主过程中经历复杂的遗传重组与进化,该现象增加了院感防控的复杂性和挑战。同时,本研究还发现1例肠道定植菌株与血流感染菌株分属不同克隆型的特殊菌株,该患者同期痰标本中分离出与感染株同克隆型菌株,表明肠道外其他部位定植同样可引发侵袭性感染,强调针对高危人群加强多部位筛查的重要性,以完善综合防控体系。
本研究存在一定局限:(1)符合纳入标准的患者较少,仅获得6对肠道定植菌株和血流感染菌株,限制了结论的推广;(2)主要聚焦肠道定植菌株和血流感染菌株的临床流行病学与基因组特征关联性,缺乏动物模型对该结果进行验证,亦缺乏CRKP从定植至血流感染机制的深入探讨。
综上,本研究结合临床资料与基因组学分析,证实了CRKP肠道定植菌株与血流感染菌株的遗传关系,明确了肠道定植菌株为血流感染的直接来源,为肠道定植的干预策略制定及经验用药优化提供了重要理论依据。CRKP肠道定植继发血流感染死亡率较高,院感部门应加强重点科室的管理,科学制定院内感染防控方案。鉴于本研究存在的局限性,未来研究将继续跟踪CRKP定植情况,积极收集更多肠道定植与感染菌株组配对样本,采用多维度方法,系统解析CRKP肠道定植继发感染的传播机制,并基于全基因组测序技术结合机器学习构建精准预警模型,强化CRKP定植及感染患者的筛查和随访,提升医疗机构多部门协作开展实时监测,有效阻断耐药菌在院内的传播链条,从而实现精准干预、及时预警、个体化治疗,以降低CRKP感染发生率,提升患者安全与医疗质量。
参考文献
[1]Jean S S, Harnod D, Hsueh P R. Global threat of carbapenem-resistant gram-negative bacteria[J]. Front Cell Infect Microbiol, 2022, 12: 823684.
[2]Dong L T, Espinoza H V, Espinoza J L. Emerging superbugs: the threat of carbapenem resistant enterobacteriaceae[J]. AIMS Microbiol, 2020, 6(3): 176-182.
[3]Ge X, Zhou Y, Jin H, et al. Genomic insights and antimicrobial resistance profiles of CRKP and non-CRKP isolates in a Beijing geriatric medical center: emphasizing the blaKPC-2 carrying high-risk clones and their spread[J]. Front Microbiol, 2024, 15: 1359340.
[4]孟雪斐, 张鸿娟, 宋贵波, 等. 肠道定植耐碳青霉烯类肠杆菌目细菌分子特征及其感染相关研究[J]. 中国抗生素杂志, 2023, 48(5): 563-572.
[5]Hassoun-Kheir N, Hussien K, Karram M, et al. Clinical significance and burden of carbapenem-resistant Enterobacterales (CRE) colonization acquisition in hospitalized patients[J]. Antimicrob Resist Infect Control, 2023, 12(1): 129.
[6]Baker K S, Jauneikaite E, Nunn J G, et al. Evidence review and recommendations for the implementation of genomics for antimicrobial resistance surveillance: reports from an international expert group[J]. Lancet Microbe, 2023, 4(12): e1035-e1039.
[7]Jauneikaite E, Baker K S, Nunn J G, et al. Genomics for antimicrobial resistance surveillance to support infection prevention and control in health-care facilities[J]. Lancet Microbe, 2023, 4(12): e1040-e1046.
[8]Baker K S, Jauneikaite E, Hopkins K L, et al. Genomics for public health and international surveillance of antimi-crobial resistance[J]. Lancet Microbe, 2023, 4(12): e1047-e1055.
[9]U.S. Centers for Disease Control and Prevention. Facility guidance for control of carbapenem-resistant Enterobacteriaceae (CRE)[EB/OL]. [2025-08-02]. https://www.cdc.gov/infection-control/media/pdfs/guidelines-cre-guidance-508.pdf.
[10]中华人民共和国卫生部. 医院感染诊断标准(试行)[J]. 中华医学杂志, 2001, 81(5): 314-320.
[11]CLSI. Performance standards for antimicrobial susceptibility testing: CLSI-M100[S]. Wayne, PA: Clinical and Laboratory Standards Institute, 2023.
[12]U.S. Food & Drug Administration. Antibacterial suscepti-bility test interpretive criteria[EB/OL]. (2025-04-14)[2025-08-02]. https://www.fda.gov/drugs/development-resources/antibacterial-susceptibility-test-interpretive-cri-teria.
[13]Lam M M C, Wick R R, Watts S C, et al. A genomic surveillance framework and genotyping tool for Klebsiella pneumoniae and its related species complex[J]. Nat Commun, 2021, 12(1): 4188.
[14]Temkin E, Solter E, Lugassy C, et al. The natural history of carbapenemase-producing enterobacterales: progression from carriage of various carbapenemases to bloodstream infection[J]. Clin Infect Dis, 2024, 79(1): 22-29.
[15]刘志盈, 陈云波, 嵇金如, 等. 2022年全国血流感染细菌耐药监测报告: 革兰阴性菌[J]. 中华临床感染病杂志, 2024, 17(1): 42-57.
[16]Soares de Moraes L, Gomes Magalhaes G L, Material Soncini J G, et al. High mortality from carbapenem-resistant Klebsiella pneumoniae bloodstream infection[J]. Microb Pathog, 2022, 167: 105519.
[17]Holmes C L, Albin O R, Mobley H L T, et al. Bloodstream infections: mechanisms of pathogenesis and opportunities for intervention[J]. Nat Rev Microbiol, 2025, 23(4): 210-224.
[18]de Souza I L A, Cappellano P, Ferreira D B, et al. Carbapenem-resistant Klebsiella pneumoniae bloodstream infections in haematological malignances and hematopoietic stem cell transplantation: clinical impact of combination therapy in a 10-year Brazilian cohort[J]. PLoS One, 2024, 19(1): e0297161.
[19]Şimşek Bozok T,Bozok T,Şahinoglu M S,et al. Bloodstream infections caused by carbapenem-resistant Klebsiella pneumoniae: analysis of risk factors, treatment responses and mortality[J]. Infect Dis (Lond), 2025, 57(4): 350-360.
[20]Zhang F S, Li L Y, Zhao Y X, et al. Molecular characterization of hybrid virulence plasmids in ST11-KL64 KPC-2-producing multidrug-resistant hypervirulent Klebsiella pneumoniae from China[J]. Front Microbiol, 2024, 15: 1353849.
[21]Wang J N, Feng Y, Zong Z Y. Worldwide transmission of ST11-KL64 carbapenem-resistant Klebsiella pneumoniae: an analysis of publicly available genomes[J]. mSphere, 2023, 8(4): e0017323.
[22]Henoun Loukili N, Perrin A, Gaillot O, et al. Is intestinal colonization with multidrug-resistant Enterobacterales associated with higher rates of nosocomial Enterobacterales bloodstream infections?[J]. Int J Infect Dis, 2025, 150: 107274.
[23]Hu H D, Wang Y P, Sun J, et al. Risk factors and molecular epidemiology of intestinal colonization by carbapenem-resistant Gram-negative bacteria in patients with hematological diseases: a multicenter case-control study[J]. Microbiol Spectr, 2024, 12(7): e0429923.
[24]倪晓平. 从南丁格尔到AI时代: 医疗保健环境清洁的百年演进与未来展望[J]. 中国感染控制杂志, 2025, 24(3): 295-300.
- 搜索
-
- 1000℃李寰:先心病肺动脉高压能根治吗?
- 1000℃除了吃药,骨质疏松还能如何治疗?
- 1000℃抱孩子谁不会呢?保护脊柱的抱孩子姿势了解一下
- 1000℃妇科检查有哪些项目?
- 1000℃妇科检查前应做哪些准备?
- 1000℃女性莫名烦躁—不好惹的黄体期
- 1000℃会影响患者智力的癫痫病
- 1000℃治女性盆腔炎的费用是多少?
- 标签列表
-
- 星座 (702)
- 孩子 (526)
- 恋爱 (505)
- 婴儿车 (390)
- 宝宝 (328)
- 狮子座 (313)
- 金牛座 (313)
- 摩羯座 (302)
- 白羊座 (301)
- 天蝎座 (294)
- 巨蟹座 (289)
- 双子座 (289)
- 处女座 (285)
- 天秤座 (276)
- 双鱼座 (268)
- 婴儿 (265)
- 水瓶座 (260)
- 射手座 (239)
- 不完美妈妈 (173)
- 跳槽那些事儿 (168)
- baby (140)
- 女婴 (132)
- 生肖 (129)
- 女儿 (129)
- 民警 (127)
- 狮子 (105)
- NBA (101)
- 家长 (97)
- 怀孕 (95)
- 儿童 (93)
- 交警 (89)
- 孕妇 (77)
- 儿子 (75)
- Angelababy (74)
- 父母 (74)
- 幼儿园 (73)
- 医院 (69)
- 童车 (66)
- 女子 (60)
- 郑州 (58)